Реверсинг работы кода ДНК и клеточных механизмов
Рисунок: Микроскопическое изображение клеток (стрянка толстой кишки, окрашено гематоксилином–эозином).
Структура клетки и информационных систем: метафора и перспектива
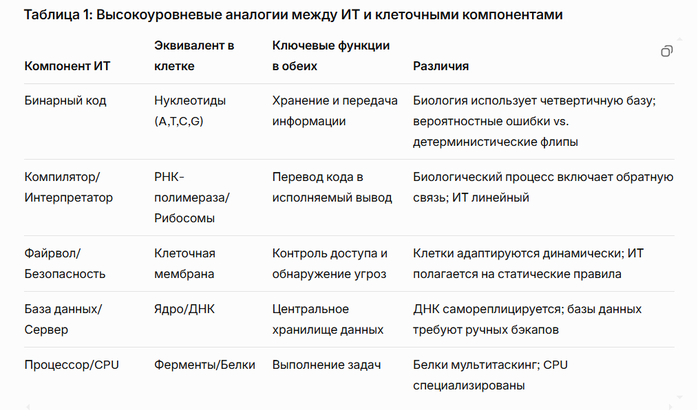
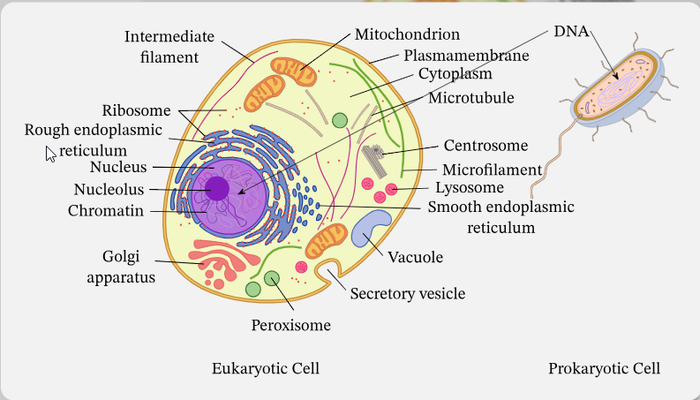
Клетка – высокоорганизованная система со множеством подсистем (ядро, митохондрии, рибосомы и др.), каждая из которых выполняет определённые функции. Это похоже на компьютерную систему, где процессор, память, устройства ввода-вывода и сети формируют аппаратное обеспечение. В биологии ДНК хранит «инструкции» сборки белков, аналогично тому, как в компьютере инструкции хранятся в двоичном коде. Как и машинный код, трансляция «программы жизни» контролируется «компилятором» (рибосомой), читающим «код» мРНК. Рибосомы «читают» генетический код и собирают белки подобно тому, как на заводе по чертежу собирают механизм[1]. Таким образом, ДНК можно рассматривать как «чертёж» или «программу» организма, а операции транскрипции/трансляции – как компиляцию и выполнение этого кода.
В информатике, подобно биологии, широко используются аналогии «вирус-компьютер». Термин «компьютерный вирус» был введён Леонардом Адлеманом (первопроходцем ДНК-вычислений): он заметил сходство между самовоспроизводящимися программами и биологическими вирусами[2]. Однако при этом системы защиты у биологического организма и у компьютера сильно различаются: иммунная система человека – «умнейший механизм», способный учиться на прошлых атаках и защищать организм от всё новых патогенов, тогда как современные антивирусы и фаерволы далеки от такой эффективности[3]. Ученые отмечают, что было бы полезно перенять принципы иммунной системы для киберзащиты, но «механизмы живых организмов куда более сложны»[4]. Это подчёркивает: хотя аналоги есть, компьютер и биология развивались разными путями, и прямое отождествление их компонентов может вести к упрощениям. Тем не менее, рассуждения о клетке как о «биологическом компьютере» помогают мыслить абстрактно – например, рассматривать мутацию ДНК как «сбой программы» (с ошибкой в коде) и болезни как «эксплойты» в системе.
Уровни абстракции: от кода к архитектуре
В программировании понятия высокого уровня (исходный код) и низкого уровня (машинные инструкции) являются основными. Аналогично, в биологии ДНК (или геном) – это «исходный текст» на четырёхбуквенном алфавите (A, T, C, G), который транскрибируется в РНК и «выполняется» рибосомами, выстраивая цепочку аминокислот в белке. Эти этапы можно сопоставить с компиляцией и выполнением кода. Есть и дополнительные уровни: эпигенетическая регуляция (метилирование, модификации гистонов) действует как «параметры конфигурации» или механизмы авторизации, управляя тем, какие гены «включены», подобно переключению флагов в системе. Ошибки в этой «системе» ведут к «сбойным процессам» – например, к неправильно свернутым белкам или неконтролируемому делению клеток (раку), аналогично тому, как сбой в прошивке вызывает отказ техники. Однако, в отличие от цифровых систем, биологическая «вычислительная машина» гибка: в ней присутствуют обратные связи, шумовые эффекты и стохастичность, что делает её устойчивой, но и непредсказуемой.
Сложность клеточных систем и моделирование
Работа клетки происходит на множестве масштабов: от атомно-молекулярного (размеры Å) до межклеточного (тканевые связи) и временных (фемтосекунды до часов/дней). Поэтому для полного понимания нужен мультиуровневый подход. Современные экспериментальные методы (кристаллография, МД-томография, суперштативная микроскопия, секвенирование) дают лишь фрагменты картины. Вычислительные методы вступают на «поле битвы» как «компьютерный микроскоп». Например, молекулярная динамика (МД) сегодня моделирует системы с сотнями миллионов атомов, включая фрагменты клеток: от фотосинтетических комплексов до вирусов в каплях воды[5]. В перспективе уже предложено моделировать целую клетку – были выстроены интегративные МД-модели минимального синтетического организма JCVI-syn3A во всей сложности[6]. Это первый шаг к тому, чтобы «опросить клетку» («interrogate») на атомном уровне. С другой стороны, классические системные модели (геномно-масштабные сети реакций, кинетические модели) уже позволяют описывать метаболизм и регуляцию, но упрощают геометрию.
Чтобы «собрать слона», учёные комбинируют эти подходы («cellPACK» для статических моделей клеток, Lattice Microbes и др.). Но интеграция требует огромного объёма данных: построение реалистичной модели целой клетки требует «интегративного моделирования», основанного на совокупности разнородных экспериментальных данных (структур, концентраций, стехиометрии)[7]. Проблема в том, что получить такие данные в реальном времени с высоким разрешением крайне сложно. По мере развития методов можно надеяться, что цифровые модели (MD, стохастические симуляторы, гибридные модели) научатся охватывать всё большие «куски» клетки и связывать их в единую картину.
Роль ИИ и больших данных в биологических системах
Параллельно с ростом объёма биоданных (секвенирование, протеомика, структурная биология) возрастают возможности ИТ-анализа. Искусственный интеллект уже начинает играть роль «суперкомпилятора» биологических моделей. Например, при генном редактировании CRISPR-гайды подбирают с помощью нейросетей (DeepCRISPR, CRISTA и др.)[8]. Обзор последних лет подчеркивает, что «интеграция ИИ и технологий редактирования генома открывает новые возможности для генетики, биомедицины и здравоохранения с существенным влиянием на здоровье человека»[9].
ИИ-модели уже используются для точного предсказания эффективности генно-редактирования: например, алгоритм SPROUT на обучении на миллионах событий CRISPR-Cas9 смог с высокой точностью прогнозировать результат ремонта ДНК в T-клетках[10]. В то же время ИИ применяют к анализу «мультиомики» – больших наборов данных транскриптомики, протеомики и пр.
Авторы последних исследований отмечают: с появлением мегаданных о генах, РНК, белках и метаболитах ИИ становится необходимым для извлечения смыслов[11][10]. Пример: в онкологии ИИ-кластеризация помогает выделить подтипы опухолей по геному, а CRISPR-редактирование может «отключать» онкогены или перепрограммировать иммунные клетки для атаки конкретных мутантных клеток[10]. В «печеночно-виртуальной» модели клеток ИИ уже способен интегрировать данные из разных органов (печени, почек) и предсказывать тканеспецифические реакции на лекарство[12] (например, токсичность в клетках печени соответствует клиническим результатам).
Быстрый прогресс ИИ в биологии иллюстрируют прорывы вроде AlphaFold 2: нейросеть DeepMind научилась предсказывать трехмерную структуру белков почти с точностью эксперимента (погрешность ~1 Å), значительно опережая предыдущие методы[13]. Это открывает путь к быстрому конструированию моделей белков и их комплексов, что ускоряет дизайн препаратов и понимание клеточных машин. Аналогично технологии «глубокого обучения» помогают анализировать медицинские данные (изображения, ЭЭГ, томограммы), связывать геномные варианты с болезнями и «переводить» биохимию в понятие вычислений.
Инструменты и ускорение исследований
Для достижения долгосрочных целей нужны мощные инструменты и инфраструктура. Ученые предлагают масштабные проекты наподобие «Искусственной виртуальной клетки» (AI-virtual cell)[14][15]. Такая «виртуальная клетка» создала бы единую модель, объединяющую химические, электрические и механические процессы; её можно использовать для ** in silico ** экспериментов вместо in vivo[16]. Как отмечают исследователи Стэнфорда, «AI позволяет учиться напрямую на данных и обнаруживать возникающие свойства сложных биологических систем»[17].
Глобальный проект требует объединения разведок: уже сейчас в распоряжении науки петабайты секвенирования – хранилище Short Read Archive NIH насчитывает >14 PB данных (примерно в 1000 раз больше, чем датасет для обучения ChatGPT!)[18]. Необходимо инвестировать в вычислительную мощь (HPC, GPU-кластеры, квантовые компьютеры) и инструменты передачи знаний (стандарты SBML/CellML для моделей, базы данных генов и белков, платформы для данных мультиомикса).
Квантовые вычисления рассматриваются как наступающий рубеж в биофармакологии: гибридные квантово-классические алгоритмы позволяют более эффективно моделировать химические взаимодействия в лекарственных молекулах и ускорять оптимизацию молекулярных свойств[19]. Хотя это область ранней стадии, уже сегодня в литературе отмечено: «квантовые компьютеры предлагают новые подходы для ускорения открытия лекарств за счёт улучшенного моделирования молекул, оптимизации и обеспечения безопасности данных»[19].
Таким образом, чтобы за 10–20 лет освоить борьбу с генетическими болезнями, нужны как экспериментальные, так и ИТ-технологии: редактирование генома (CRISPR/Base-редакторы), синтетическая биология (конструирование биологических «программных модулей»), виртуальное тестирование лекарств (атомистические и клеточные симуляции), высокопроизводительные вычисления и сквозные стандарты обмена данными.
Уже ведутся международные усилия: например, Chan Zuckerberg Initiative строит платформы для обмена биологическими ИИ-моделями и наборами данных. Большое внимание уделяется интеграции ИТ-безопасности: генетические данные пациентов требуют надёжного шифрования и приватности, а наработки киберзащиты можно (по аналогии с иммунитетом) адаптировать для биомедицины.
Конкретные шаги и перспективные задачи
Ниже приведён примерный план – от фундаментальных задач к практическим, чтобы вскрыть «низкий уровень» работы клетки через призму ИТ:
Декодирование и систематизация биологической информации: продолжать полную расшифровку генома, транскриптомов и протеомов у разных клеточных типов и состояний. Разрабатывать семантические онтологии и базы знаний (например, глобальные базы “ген – заболевание”), аналогичные реестрам программных компонентов. Автоматизация анализа последовательностей (как компилятор), оптимизация алгоритмов выравнивания и аннотации (BLAST/GPU-ускорение) – эти задачи ближе к «низкоуровневому программированию» в биологии.
Многомасштабное моделирование клетки: наращивать возможности вычислений от отдельных молекулярных взаимодействий до моделей целой клетки. Этапы: 1) кинетические модели отдельных путей (как малые фрагменты кода), 2) статические 3D-модели органелл (аналог аппаратных схем), 3) динамические модели всех метаболических и сигнальных сетей. Использовать платформы (Virtual Cell, COPASI, Lattice Microbes) для структурных и химических симуляций, а также новые подходы (GPU, FPGA, распределённые вычисления).
Развитие искусственного интеллекта и больших данных: строить комплексные модели на основе глубинного обучения, объединяя «омикс»-данные, изображения и текстовые медицинские данные. Обучать нейросети на скрин-системах CRISPR, данных РНК-секвенирования и протеомов, чтобы предсказывать паттерны экспрессии, взаимодействия белков, эффективность лекарств. Создавать «виртуальную клетку» – обученную сеть, которая может моделировать состояние клетки после любых генетических вмешательств[17]. Применять технологии генеративного ИИ (GraphGPT, ProteinGAN) для синтеза новых биохимических путей и молекулярных структур.
Синтетическая биология и инженерия клеток: проектировать и конструировать искусственные генетические схемы по принципам разработки ПО. Клетка рассматривается как хост для «биокирпичиков» (BioBricks), и им можно придать функции аналогично модулям ПО. Исследования уже предлагают применять шаблоны программной инженерии: например, выделять паттерны и модельные языки для «модульных» частей ДНК и проверки их безопасности[20]. Такая инженерия позволит «записывать» нужные алгоритмы в геном и строить клетки с заданными функциями (чистка токсинов, производство лекарств, восстановление тканей).
Иммунная и противовирусная инженерия: рассматривать иммунную систему как ИТ-модель «самозащиты». Разрабатывать методы программирования иммунных клеток (CAR-T, TCR-аналоги) для борьбы с раком и инфекциями, используя аналогии с построением надежных распределённых систем. «Вирусы» организма можно изучать как «зловредное ПО»: обратная разработка механизмов атак (например, как вирус РНК вводит инструкции в клетку) поможет создать новые антибиотики и вакцины. С другой стороны, учёные предлагают перенимать стратегии иммунитета для создания самоблокирующихся «антивирусных» алгоритмов в IT.
Инфраструктура и стандартизация: реализовывать совместные платформы для обмена данными и моделями, подобно тому, как открытые репозитории кода (GitHub) трансформировали софт. Развивать облачные биоплатформы (например, CZI AI Cell Models) и федеративное обучение, чтобы собирать разрозненные данные (болезни, геномы, лабораторные экспонаты) без нарушения конфиденциальности. Разрабатывать открытые стандарты моделирования (SBML, CellML, ONNX для биомоделей) и ресурсы для совместной валидации. Всё это ускорит «DevOps биологии» – непрерывную интеграцию эксперимента и симуляции[21].
Междисциплинарное образование: готовить новые кадры на стыке ИТ, биологии и медицины. Это лабораторно-инженерные кадры, которые понимают микромолекулярную биологию и в то же время могут писать код, проектировать алгоритмы и настраивать суперкомпьютеры. Хакатоны по анализу геномных данных, совместные курсы биоинформатики и кибербезопасности, двойные PhD — всё это ускорит «конвергенцию» дисциплин.
Примеры приложений и прорывные технологии
Редактирование генома и генные терапии: уже ведутся клинические испытания CRISPR/Cas-терапий при серповидноклеточной анемии, β-талассемии и некоторых раках. Глубокое понимание работы клеточных «инструкций» позволило создать системные платформы, где ИИ подбирает точные генные «патчи». Успехи недалёкого будущего – лечение ранее неизлечимых генетических болезней (мутации единственной нуклеотидной подстановки), ретинальных дистрофий, модификация иммунных клеток против рака.
Виртуальные испытания препаратов: «Вычислительные клетки» (наброски моделей тканей и органов) позволят массово прогнозировать токсичность и эффективность лекарств до клинических испытаний. Уже сейчас AI-модели клеток демонстрируют, что по мультиомическим данным можно предсказать реакцию тканей на лекарство[22], что облегчит поиск новых лекарственных мишеней и ускорит вывод препаратов на рынок.
Иммунные и онкогенные симуляции: цифровые модели помогут смоделировать, как именно мутации превращают здоровую клетку в злокачественную, и предсказывать ответ иммунитета. Генно-инженерные клетки (CAR-T) можно «протестировать» на цифровых двойниках пациентов (virtual twins) – так же как инженеры тестируют программное обеспечение на виртуальных машинах[23]. Это ускорит персонализированное лечение: врачи смогут подобрать терапию, смоделировав её действие в «цифровом организме» пациента[23].
Быстрое протезирование и регенерация: ИТ-технологии объединят биопринтинг тканей и носимые сенсоры. Например, созданы «искусственные органы» на 3D-принтере (биофабрикация) и чипы-тканесимуляторы (organs-on-chip). Облачные вычисления будут обрабатывать данные о состоянии тканей пациента в реальном времени (аналог IoT в промышленности). Это даст возможность скорей восстановить сильно повреждённые ткани (печень, сердце) – своего рода «горячий перезапуск» органа.
Перспективные направления (5–10 лет)
Ниже 10 ключевых направлений развития исследований (ранжированы по приоритету/срочности):
AI-виртуальная клетка и мультиомные модели (очень высокая срочность): завершение создания виртуальной клетки (Stanford, CZI) позволит глубже понимать болезни и тестировать терапии in silico[17][23]. Это комплексная цель, требующая глобального коллаборативного проекта. Успех в ней приведёт к новому этапу биомедицины, сравнимому с аттестатом Человеческого генома[15].
Интеграция больших данных и ИИ в биомедицине (высокая срочность): объединить геномные, протеомные, изображенческие и клинические данные с помощью глубокого обучения. Усовершенствовать алгоритмы интерпретации мутаций, предсказания функций генов и отбора лекарств[11][24]. Развивать «глубокое обучение по биоданных» (single-cell, spatial omics) и generative AI для синтеза биологических карт.
Продвинутое генное редактирование и терапия (высокая срочность): совершенствовать методы CRISPR (базовое и prime-редактирование) с минимизацией побочных эффектов. Интегрировать ИИ-поддержку для оптимизации генно-терапевтических вмешательств[8][11]. Сосредоточиться на проблемах неизлечимых заболеваний: рак (исключение онкогенов), вражденные мутации (лечением основную причину), вирусы (устойчивые к лекарствам – их «неактивизация» генетическими методами).
Системная биология и кинетика клеточных сетей (средняя–высокая срочность): развитие математических моделей всего клеточного цикла и тканевой организации. Использовать стохастические симуляции для шумовых эффектов, гибридные детерминистические модели для обмена веществ. Совмещать модели сигналинга, метаболизма и механики (multi-scale). Это базовый фундамент для «отладки» клеточной работы, как отладка ПО.
Синтетическая биология и «биоинженерия» (средняя срочность): создавать универсальные биомодули (BioBricks) по аналогии с программными библиотеками. Это позволит перестраивать клетки «с нуля», внедрять новые функции (биопроизводство лекарств, регенерация тканей). Ускорить стандартизацию: обеспечить библиотеку безопасных генетических «скриптов» и методов их компиляции (сборки). Это откроет двери к созданию искусственных «программируемых» клеток.
Виртуальное тестирование лекарств и терапии (высокая срочность): развитие платформ для «in silico» скрининга лекарств на базе биомоделей[22]. Интегрировать кинетические и структурные модели для оценки новых соединений. ИИ-ускоренный докинг, химическое «начертание» препаратов, объяснимые модели токсичности. Уменьшить зависимость от животных испытаний – виртуальные модели уже демонстрируют предсказание тканевых ответов[22].
Иммунная инженерия и противораковая биология (высокая срочность): перевод иммунологии в ИТ-парадигму: моделирование иммунных клеток, редактирование T-клеток и NK-клеток с помощью CRISPR (программисты собственной иммунной системы). Аналогично киберзащите, создавать адаптивные «биосистемы защиты» от рака и инфекций. В рамках COVID-19 и рака такие исследования уже ведутся, но их масштаб нужно увеличить для всех типов опухолей и вирусов.
Инфраструктура HPC и квантовые вычисления в биологии (средняя срочность): наращивать суперкомпьютерные и квантовые мощности для биосимуляций. Создавать биоинформационные «облака» для совмещения усилий (подобно CERN Grid в физике). Инвестировать в разработку специализированных процессоров (биоцели, акселераторы нейронных сетей для биомоделей). Квантовые алгоритмы могут дать крутой прирост в симуляции молекул и оптимизации клинических данных[19].
Глобальное сотрудничество и открытые данные (средняя срочность): координировать мировую науку, как мы объединились в ряде мегапроектов (Human Cell Atlas, Cancer Moonshot). Создавать открытые реестры пациентов с геномной информацией (безопасные, анонимные) для совместного анализа. Подобно открытым исходникам в IT, продвигать доступность моделей и симуляций (уверенность что результаты воспроизводимы и проверяемы всеми).
Образование и подготовка специалистов (очень высокая срочность): реализовать образовательные программы на стыке ИТ, биологии и медицины. Подготовить bioinformaticians, биоинженеров и медицинских физиков, способных «читать» как генетический код, так и машинный код. Только образованное междисциплинарное сообщество сможет реализовать стратегию «от кода к клетке» и воплотить перечисленные направления за ближайшее десятилетие.
Эти направления рассчитаны на 5–10 лет. Успех в них – от создания виртуальных клеток до освоения ИИ и нового железа – определит, сможем ли мы в этом сроке лечить болезни, вызванные повреждением генома (рак, наследственные мутации, тяжелые повреждения тканей). Все они опираются на синтез биологии и ИТ: изучение «низкого уровня» клеток, как отладка и проектирование сложных систем. Мы стоим на пороге биотехнологической революции, где знание принципов работы компьютеров и ПО может дать ключ к пониманию и модификации «ПО жизни».