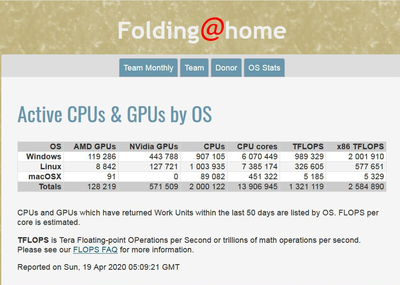
Обновление Folding@Home SARS-CoV-2 (10 марта 2020 г.)
Обновление Folding@Home SARS-CoV-2 (10 марта 2020 г.)
Это обновленная информация об усилиях Folding@Home по оказанию помощи исследователям во всем мире в борьбе с COVID-19.
После начального контроля качества и ограниченных этапов тестирования команда Folding@Home выпустила начальную волну проектов, имитирующих белки-мишени с потенциальной лекарственной активностью SARS-CoV-2 (вирус, вызывающий COVID-19) и связанный с ним вирус SARS-CoV (для которого доступно больше структурных данных) в полном объеме на Folding@Home. Большое спасибо большому количеству участников Folding@Home, которые до сих пор помогали нам, работая в режимах бета-версий или в расширенном режиме.
Эта начальная волна проектов направлена на лучшее понимание того, как эти коронавирусы взаимодействуют с человеческим рецептором ACE2, необходимым для проникновения вируса в клетки-хозяева человека, и как исследователи могли бы вмешиваться в них посредством создания новых терапевтических антител или небольших молекул, которые могут нарушать их взаимодействие.
В ближайшие дни мы надеемся воспользоваться некоторыми из новых структурных биологических и биохимических данных, которые быстро публикуются исследователями по всему миру, которые работают над пониманием этих вирусов и стратегий по их победе. Эта работа была в основном распространена на серверах предпечати, таких как bioRxiv и chemRxiv, которые направлены на то, чтобы сделать исследования быстро доступными как для других исследователей, так и общественности для других ученых, чтобы они могли широко оценить и немедленно начать опираться на них. Мы также наладили несколько новых сотрудничества с другими лабораториями, где, как мы надеемся, Folding@Home окажет ценную поддержку в исследованиях COVID-19.
В то время как мы быстро выпустим наборы данных моделирования для использования или анализа другими, мы стремимся искать альтернативные конформации и скрытые карманы внутри наиболее многообещающих лекарственных целей, которые можно увидеть только при моделировании, а не в статических рентгеновских структурах. Мы надеемся, что эти структуры - однажды подтвержденные новыми сложными данными скрининга - могли бы помочь направить кампании виртуального скрининга или нацеливание на новые карманы, для которых еще не были доступны атомистические структуры.
Ниже мы приводим краткие описания проектов. Обратите внимание, что все входные файлы доступны на GitHub здесь, чтобы другие исследователи могли воспользоваться их преимуществами: https://github.com/foldingathome/coronavirus
Этот репозиторий будет развиваться в ближайшие дни по мере добавления новых проектов и документации. Мы начнем публиковать наборы данных со структурами на общедоступных серверах, как только у нас появятся полезные данные для отчета.
Во всех проектах используется новое ускоренное ядро Core22, основанное на движке биомолекулярного моделирования OpenMM с открытым исходным кодом.
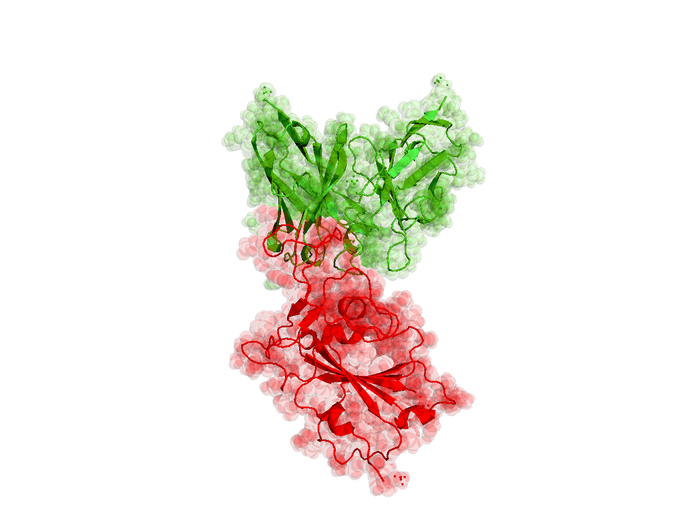
Домен RBD SARS-CoV-2 в комплексе с человеческим рецептором ACE2 (PDBID: 6vsb, 6acg) [http://dx.doi.org/10.1126/science.abb2507, http://dx.doi.org/10.1371/journal.ppat.1007236]
Проект 11741: Домен связывания рецептора коронавируса SARS-CoV-2 (вызывающий COVID-19) в комплексе с человеческим рецептором ACE2. атомы: 165550, очки: 15396
Проект 11746: Домен связывания рецептора коронавируса SARS-CoV-2 (вызывающий COVID-19) в комплексе с человеческим рецептором ACE2 (структура, альтернативная 11741). атомы: 182699, очки: 16615
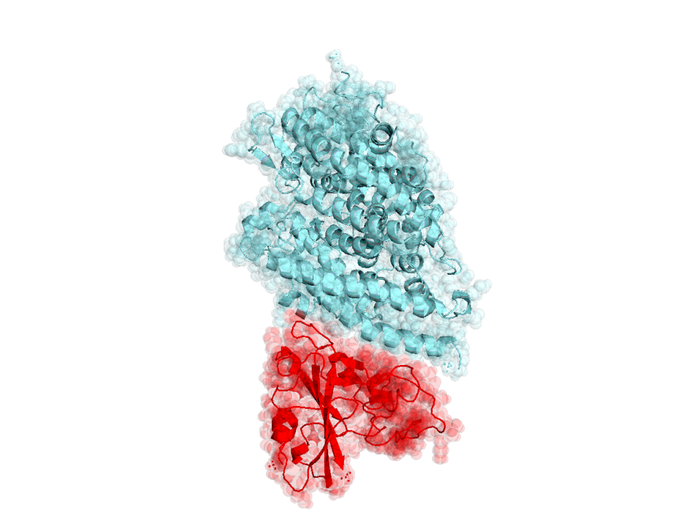
Основная протеаза SARS-CoV-2 в комплексе с ингибитором N3 (PDBID: 6lu7) [Пока не опубликовано]
Проект 11742: Коронавирусная протеаза SARS-CoV-2 (вызывающая COVID-19) протеазы в комплексе с ингибитором. атомы: 62227, очки: 9405
Проект 11743: Коронавирусная протеаза SARS-CoV-2 (вирус, вызывающий COVID-19) - потенциальная лекарственная мишень. атомы: 62180, очки: 9405
Домен RBD SARS-CoV-2 в комплексе с Fab-фрагментом нейтрализующего антитела S230 человека (PDBID: 6nb7, 6nb8, 2ghv) http://dx.doi.org/10.1016/j.cell.2018.12.028 (как для 6nb7, так и 6nb8), http://dx.doi.org/10.1074/jbc.M603275200
Проект 11744: Домен связывания с рецептором коронавируса SARS-CoV (вирус, вызывающий SARS), захваченный антителом SARS-CoV S230. атомы: 109578, очки: 7608
Проект 11745: Домен связывания рецептора SARS-CoV (вызывающий вирус SARS) коронавируса, мутировавший в SARS-CoV-2 (вирус, вызывающий COVID-19), захваченный антителом SARS-CoV S230. атомы: 110370, очки: 7685
Чтобы связаться с нами для обсуждения сотрудничества или данных, пожалуйста, напишите нам по адресу: foldathome@choderalab.org
Особая благодарность аспиранту TBCP Rafal Wiewiora и аспиранту CBM Ivy Zhang за их работу по моделированию этих структур на основе существующих экспериментальных данных и подготовке этих проектов, а также всем участникам Folding@Home, которые помогают сделать эту работу возможной!
~ Команда Chodera lab SARS-CoV-2 и Консорциум Folding @ home ~
Хотите принять участие в распределенных вычислениях, тогда, Вам сюда:
Желание
Мой проект на python
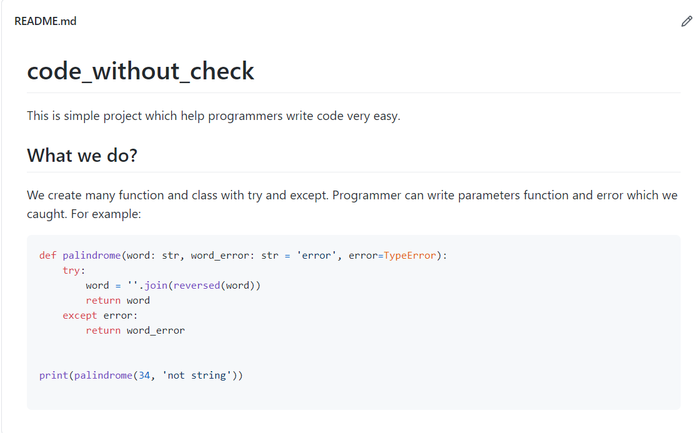
Весь код ЗДЕСЬ
Решил я создать свою библиотеку и рассказать про неё вам. Хочется мне, чтобы вы её оценили и сказали как вам сама задумка и надо ли её продолжать.
Идея в том, чтобы упростить жизнь разработчикам и уменьшить код на python. Представьте что вам не нужно писать try и except для проверки какой-либ функции. Вы просто передаете обычные параметры функции, пишите что выведется при ошибки ( в блоке except ) и какую ошибку вы будете ловить. Я кое-как рассказал об этом в README.md
Я пока точно не определился в каком направление будут создаваться функции, но мне кажется что лучше начать с самых базовых операций, потому что они используются чаще всего. Также вы можете поделиться своим мнением в комментариях.
Этот пост был нестандартным. Я хотел поделиться с вами мои мини проектом.
Нужна обратная связь по моему творению
Всем доброго дня, рад что вы обратили на этот пост своё внимание!
Свой рассказ я начну с небольшой предыстории:
С недавних пор, я создал себе свой сайтик, сразу скажу не я его писал, я всего ли немножко его доработал. Причина моих действий стала моя скука и ощущение умственной деградации.
Решил так сказать проверить свой скилл в понимании CSS и HTML!
Ну и вот что из этого вышло -
ОРИГИНАЛ -
РЕЗУЛЬТАТ -
Я знаю, что это ДАЛЕКО не идеал! Поэтому мне было бы полезно послушать вашу критику со стороны. И к слову всё это хостится на GitHub Pages, может кому-то будет полезно!
Вот ссылка на сам сайтик : alekszavg.github.io
Вот ссылка на репозиторий этого сайта : github.com/AleksZavg/alekszavg.github.io
Что вы можете мне посоветовать?
Самый лёгкий и простой кроссплатформенный медиаплеер
Обзор
бесплатный (как в Freedom) медиаплеер для командной строки. Он поддерживает широкий спектр форматов медиафайлов, аудио- и видеокодеков, а также типов субтитров.
Сценарии
Мощные возможности создания сценариев могут заставить игрока делать практически все. В вики есть большой выбор пользовательских скриптов .
Вывод видео высокого качества
mpv имеет видеовыход на основе OpenGL, Vulkan и D3D11, который поддерживает многие функции, любимые видеофилами, например масштабирование видео с помощью популярных высококачественных алгоритмов, управление цветом, синхронизацию кадров, интерполяцию, HDR и многое другое.
Экранный контроллер
стремится к минимализму и не предоставляет реального графического интерфейса, он имеет небольшой контроллер поверх видео для базового управления.
Декодирование видео на GPU
может использовать большинство API аппаратного декодирования на всех платформах. Аппаратное декодирование можно включить во время выполнения по запросу.
СКРИНШОТЫ
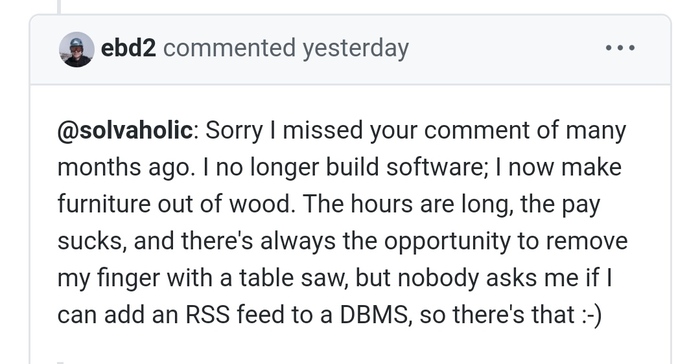
Ответ на давний Github-вопрос
Извините что я не заметил ваш комментарий много месяцев назад. Я больше не пишу код, я теперь делаю мебель из дерева. Рабочий день - долгий, зарплата - хреновая, и всегда есть риск потерять палец в циркулярной пиле, но по крайней мере, никто больше не спрашивает меня, могу ли я подключить RSS-фид к базе данных. :-)