Компьютерная структура данных впервые реализована в ДНК
Мир живого и мир машин стали ещё немного ближе друг к другу. Коллектив учёных из университета Ньюкасла продемонстрировал прототип стековой памяти на базе ДНК. Само по себе использование ДНК для хранения цифровой информации — в научном мире уже не новость, но вот динамическая структура, которая позволяла бы стирать и записывать данные и выдавать их упорядоченно, создана впервые.
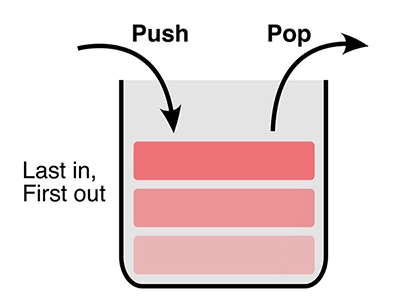
Так работает стек-память: последним пришёл — первым ушёл
Большинство ранее представленных разработок — это архивная память. На основе битовой последовательности химически синтезируется нужная цепочка ДНК, которую затем можно прочитать методом секвенирования или ПЦР. Это перспективное решение для долговременного хранения данных, которое способно обеспечить плотность записи на несколько порядков выше, чем лучшие из существующих «железных» систем. Но такой формат не предусматривает модификации данных.
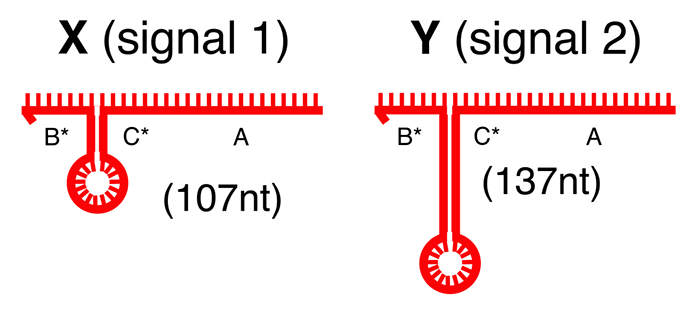
Стековая память на ДНК организована иначе. Запись и чтение информации осуществляются путём построения и усечения полимеров из одноцепочечных нитей ДНК. Биты информации кодируются при помощи двух сигнальных цепочек ДНК, одна длиной 107, а другая — 137 нуклеотидов. Далее мы будем их называть «сигнал X» и «сигнал Y».
Нуклеотиды в этих сигналах подобраны так, чтобы у них были одинаковые домены гибридизации (точки, в которых они соединяются с другими цепочками ДНК), но при этом образовывались так называемые шпильки разной длины, по которым сигналы можно было бы легко различать.
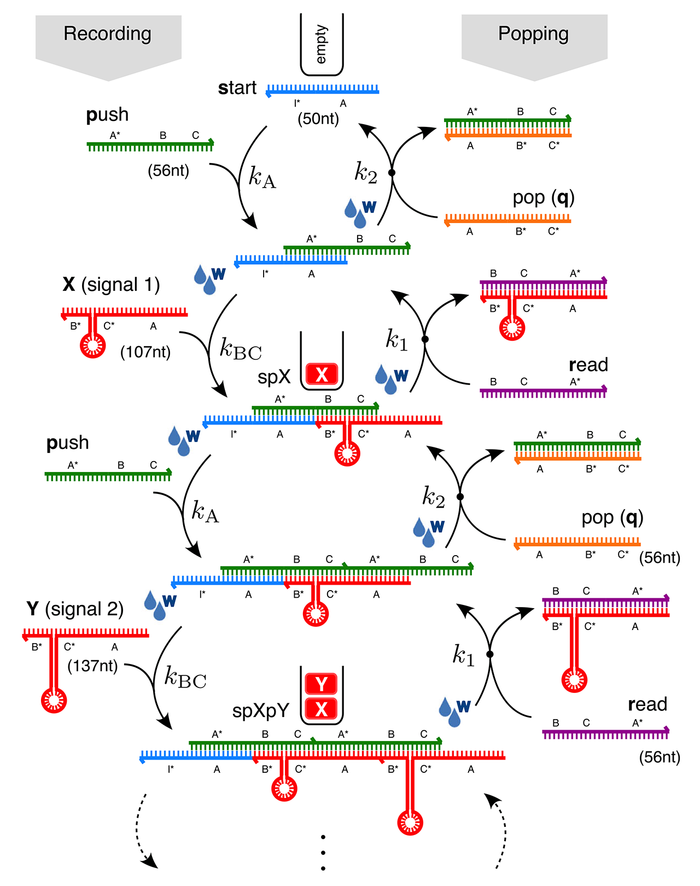
Работает всё следующим образом. В начале в раствор помещается базовая цепочка (start). Затем туда добавляется вспомогательная цепочка push, которая гибридизуется (соединяется) с цепочкой start. Получается что-то типа застёжки-молнии со свободным концом. Это делает возможным дальнейшее присоединение сигнальных цепочек, то есть запись данных. Далее в раствор добавляется сигнал X или Y, который соединяется с комплексами start-push. Добавляя «связующие звенья» push и сигналы данных, можно формировать полимеры стека сколь угодно большой длины.
Когда же информацию нужно прочитать, в раствор добавляют цепочку типа read. Она соединяется с «выступом» последнего записанного сигнала и вызывает смещение нити действующей цепочки push, гибридизованной с сигналом. Цепочка read и сигнальная цепочка образуют продукт, который отсоединяется от конца стекового полимера и высвобождается в раствор, где его можно распознать. Это эквивалентно прочтению данных. Далее в раствор можно добавить цепочку pop, которая аналогичным образом удалит крайнюю цепочку push и тем самым переместит «указатель» стека. Операции read и pop можно чередовать так, чтобы в конечном счёте укоротить стек до исходной цепочки start.
Схема простая и даже изящная, но не стоит забывать, что это очень ранний прототип технологии. То, что красиво выглядит на бумаге, далеко не так быстро и эффективно работает в пробирке. Скажем, на уравновешивание реакции после добавления новой цепочки в нынешнем варианте уходит полчаса. Причём после каждой стадии необходима механическая промывка, от тщательности которой напрямую зависит надёжность работы всей системы — иначе на поведение стека станет влиять предшествующая последовательность операций. Также стоит понимать, что одна молекула ДНК на схеме дана лишь для иллюстрации. На самом деле даже в типичном объёме 20 микролитров такой стек присутствует в количестве не менее 10 млрд копий, и их идентичность не гарантируется. Есть и ограничение на количество операций, которые ДНК способна выполнить.
Так стековые цепочки выглядят под атомно-силовым микроскопом
В общем, учёным предстоит решить ещё немало проблем. Тем не менее, опытная демонстрация технологии имеет большое значение. То, что на базе цепочек нуклеиновых кислот удалось реализовать стандартные функции, пусть даже такие простые, как push и pop, открывает перспективы не только для хранения информации, но и для вычислений на ДНК. А это значит, что сделан ещё один шаг в сторону биокомпьютеров, давно предсказанных в научной фантастике. Wetware грядёт!
С полным текстом статьи можно ознакомиться в журнале Nature Communications.