Стремительная эволюция гриппа: как одни изменения влекут за собой другие
В общественном сознании закрепилось довольно легкомысленное отношение к гриппу. Действительно, зачастую его симптомы не тяжелее простудных, да и беспокоит он нас не дольше недели, причем проходит обычно без всякого лечения. Однако история взаимодействий человека и вируса гриппа требует более серьезного подхода к этому патогену. Достаточно вспомнить, что одни из самых страшных пандемий прошлого века были вызваны этим вирусом*. Да и обычный сезонный грипп далеко не безвреден: по оценкам Всемирной организации здравоохранения, ежегодно от него и связанных с ним осложнений умирают сотни тысяч человек (в первую очередь, пожилые люди, младенцы и страдающие хроническими заболеваниями), а в годы тяжелых пандемий — миллионы. По числу унесенных жизней среди инфекционных заболеваний грипп уступает, пожалуй, только ВИЧ. Основная проблема профилактики и лечения гриппа связана с тем, что вирус очень быстро меняется, и каждый год мы имеем дело с его новыми формами, поведение которых далеко не всегда можно предсказать. Очередным шагом на пути к пониманию изменчивости вируса гриппа стал компьютерный анализ последовательностей аминокислот в белках вируса и нуклеотидов в его геноме.
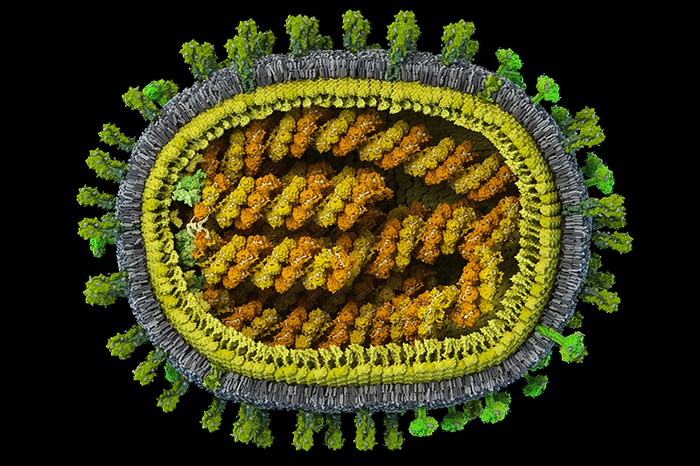
Первая в мире полная достоверная модель вируса гриппа A/H1N1 с атомным разрешением, созданная в рамках проекта Viral Park компании Visual Science при участии Национального центра биотехнологии в Мадриде. Цель проекта — построение научно достоверных 3D-моделей распространенных вирусов человека с максимальной детализацией. Специалисты Visual Science собирают воедино данные огромного количества работ по молекулярной биологии, вирусологии и кристаллографии вирусов, мнения экспертов ведущих научных центров мира и результаты молекулярного моделирования, полученные научным отделом компании. Модель в значительной степени построена на основе данных, опубликованных исследовательскими коллективами под руководством: Хуана Ортина (Испанский национальный центр биотехнологий, Мадрид, Испания), Такеши Нода (Университет Токио, Япония), Роба Ригро (Отдел взаимодействий вируса и клетки, Гренобль, Франция) и Питера Розенталя (Национальный институт медицинских исследований, Лондон, Великобритания). Точное строение генома вируса гриппа удалось смоделировать благодаря сотрудничеству с Хайме Мартин-Бенито (Испанский национальный центр биотехнологий, Мадрид, Испания), группа которого добилась уникальных результатов в описании упаковки вирусного генетического материала. Создатели модели: Иван Константинов (руководитель проекта), Юрий Стефанов (научный консультант), Анастасия Бакулина (ведущий молекулярный моделлер), Дмитрий Щербинин (молекулярный моделлер), Александр Ковалевский (3D-моделлер)
Сегментированный геном
Эволюция вирусов доставляет нам немало проблем, поскольку постоянно создает штаммы патогенов, к которым не готовы ни наш иммунитет, ни индустрия производства антивирусных препаратов и вакцин. Способность вирусных популяций быстро эволюционировать держится, с одной стороны, на огромном количестве потомства, производимого патогеном в ходе инфекции, а с другой — на высокой изменчивости вирусного генетического материала, которая дает естественному отбору шанс выявить варианты («штаммы»), противостоящие нашему иммунитету эффективнее своих предшественников. Но не все вирусы способны меняться одинаково быстро. Особенно высокая изменчивость присуща вирусам с РНК-геномом, поскольку ферменты, которые осуществляют его копирование, в отличие от полимераз, реплицирующих ДНК, не в состоянии исправлять ошибки синтеза [1]. Это приводит к появлению большего числа мутаций, чем у ДНК-вирусов.
Есть и другой механизм, позволяющий еще сильнее увеличить изменчивость. Его могут использовать РНК-вирусы, геном которых разделен на части — сегменты, напоминающие человеческие хромосомы. Если в клетку одновременно проникают два штамма одного вируса с сегментированным геномом, в потомстве может произойти перемешивание участков генетического материала — реассортация, подобно тому, как в каждом из нас перемешаны фрагменты генетического материала наших родителей. В результате реассортации возникает новый штамм вируса. Если вирусы-«родители» сильно отличались друг от друга генетически, то их потомок может приобрести набор свойств, существенно отличающих его от каждого из них. Один из патогенов, способных на такой резкий эволюционный сдвиг, — вирус гриппа. Его геном состоит из восьми отдельных молекул РНК, каждая из которых необходима для успешного осуществления жизненного цикла вируса.
Общая длина генома вируса гриппа составляет приблизительно 13 500 нуклеотидов [2]. Три самых крупных (примерно по 2300 нуклеотидов) его сегмента (PA, PB1 и PB2) кодируют вирусную полимеразу — белок, копирующий РНК и состоящий из трех крупных субъединиц. Четвертый по длине (около 1750 нуклеотидов) сегмент (HA) отвечает за синтез гемагглютинина. Этот белок заякорен в липидной оболочке вируса и отвечает за его проникновение в клетку, связываясь с рецептором на поверхности клеточной мембраны [3]. В зависимости от того, какой именно вариант гемагглютинина несет вирус, связывание может быть более или менее крепким. После этого клетка поглощает вирус, помещая его в мембранный пузырек внутри цитоплазмы. Большинство макромолекулярных комплексов, поглощаемых таким образом, перевариваются клеткой. Однако вирус избегает этой участи: его мембрана сливается с мембраной пузырька, в результате чего ее содержимое оказывается в цитоплазме. В этом процессе гемагглютинин также играет важную роль. Затем геном вируса проникает в ядро, где с него может начать считываться информация.
Сегмент размером около 1550 нуклеотидов (NP) кодирует нуклеопротеин — белок, необходимый вирусу для упаковки РНК. Множество копий такого белка распределяется по каждому из геномных сегментов, связываясь с молекулой нуклеиновой кислоты. В результате фрагменты генома образуют нуклеопротеидные тяжи, сложенные пополам и закрученные в спираль, к каждому из которых прикрепляется своя копия полимеразного комплекса [4].
Следующий сегмент длиной около 1450 нуклеотидов (NA) кодирует белок нейраминидазу. Он нужен вирусу для того, чтобы только что образовавшаяся новая частица могла «отрезать» себя от хозяйской клетки. Кстати, некоторые лекарства против гриппа (например, озельтамивир, известный под торговой маркой «Тамифлю») нацелены именно на блокирование нейраминидазы, которое позволяет снизить распространение вирусных частиц [5]. Правда, некоторые варианты нейраминидазы обеспечивают вирусу возможность избегать действия этих препаратов, делая его устойчивым к соответствующим лекарствам.
Сегмент M1/M2 длиной 1000 нуклеотидов, в соответствии со своим названием, кодирует сразу два белка — М1 и М2. Из молекул первого из них образован слой (матрикс), подстилающий вирусную липидную оболочку. Обычно М1 играет ключевую роль в формировании вирусных частиц, поскольку он взаимодействует одновременно с поверхностными белками вируса и внутренними компонентами вирусной частицы. Задача матриксного белка — собрать все составляющие воедино [6]. Белок М2 выполняет роль ионного канала. Он расположен в липидной оболочке вируса и способствует его распаковке в цитоплазме клетки [7].
Последний, самый короткий (из 865 нуклеотидов) сегмент РНК вируса гриппа отвечает за синтез двух белков, которые не попадают в зрелую вирусную частицу. Эти белки называются NS1 и NEP. Первый необходим вирусу, в частности, для того, чтобы блокировать считывание информации с клеточных молекул РНК [8]. Благодаря ему клетке приходится синтезировать преимущественно вирусные белки, оставляя свои собственные нужды. Второй белок, NEP, обеспечивает транспорт новообразованных геномных комплексов вируса из ядра к клеточной мембране, где происходит сборка вирионов [9].
Новые штаммы и поиск реассортаций
Классификация штаммов вируса гриппа основана прежде всего на том, какие именно варианты гемагглютинина и нейраминидазы входят в его состав. Широко известные комбинации букв H и N в сочетании с порядковыми номерами (например, H3N2) как раз и обозначают подтип вируса: гемагглютинин 3, нейраминидаза 2. Таких подтипов десятки, однако человека заражают лишь немногие — обычно те, у которых не слишком большие номера N и H. Наиболее давние хозяева вируса гриппа — птицы, от которых новые штаммы время от времени передаются домашнему скоту и, прямо или опосредованно, людям [10]. Чем более долгий период коэволюции провели вместе патоген и хозяин, тем менее болезненным становится их совместное существование. Птичьи штаммы вируса зачастую оказываются очень опасными после передачи новым хозяевам [11].
Известно, что именно реассортации сегментов РНК привели к возникновению штаммов, которые вызвали пандемии азиатского и гонконгского гриппа в 1957 и 1968 гг., унесшие около 2,5 млн жизней [12]. Возможно, что и испанский грипп начала прошлого века, число жертв которого шло на десятки миллионов, тоже появился в результате такой эволюционной схемы [13].
Сам процесс и последствия реассортаций изучены в настоящее время недостаточно полно. В частности, не совсем ясно, как такое резкое изменение генетического материала сказывается на дальнейшей эволюции вируса. Дело в том, что белкам, кодируемым разными сегментами вирусного генома, надо взаимодействовать между собой. Например, это важно при сборке вирусной частицы. В ходе этого процесса на поверхности зараженной клетки концентрируются вирусные белки, интегрированные в липидную оболочку. К ним относятся гемагглютинин, нейраминидаза и М2. Поверхностные белки вируса должны взаимодействовать с матриксом, который в это время формируется под клеточной мембраной. С другой стороны, М1 должен связаться с сегментами генома, чтобы они оказались упакованы в новую частицу. Да и сами сегменты должны собраться вместе таким образом, чтобы составить полный функциональный геном. Все эти ключевые для жизненного цикла вируса молекулярные взаимодействия могут быть затруднены, если белки синтезированы фрагментами геномов разных штаммов, которые еще не «притерлись» друг к другу в ходе совместной эволюции. Возможно, именно из-за этого успешные реассортации происходят сравнительно редко. Однако, коль скоро реассортация случилась, разным кускам генома, оказавшимся в одном новом штамме, надо приспособиться друг к другу.
Можно предположить, что после успешных реассортаций в геноме новой линии вируса должны закрепляться дополнительные точечные мутации, обеспечивающие такую «притирку». Эту гипотезу можно проверить биоинформатическими методами — с помощью компьютерного анализа тысяч прочитанных («отсеквенированных») геномов различных штаммов вируса, содержащихся в открытых базах данных в Интернете.
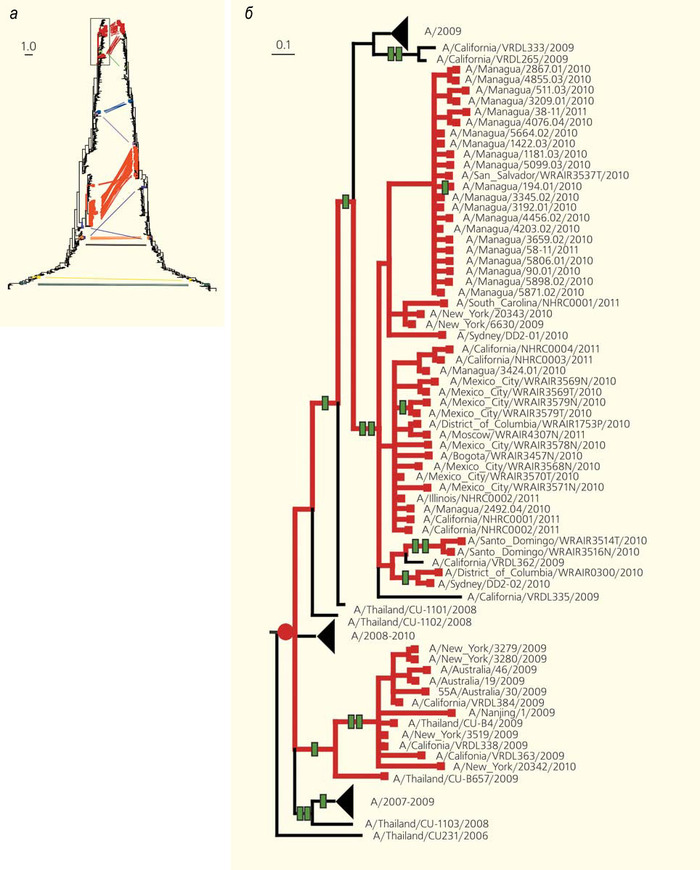
Для этого, прежде всего, необходимо узнать, что в ходе эволюции данной линии вируса гриппа произошла реассортация. Здесь на помощь приходят методы восстановления эволюционных деревьев. С помощью компьютерных программ можно сравнить ряд последовательностей одного и того же гена, взятых из большого числа особей (например, отдельных штаммов вируса), и понять, эволюционное дерево какой формы наиболее вероятно описывает историю этих штаммов. На таком дереве более похожие последовательности будут «сидеть» на близких ветках. Обычно все гены в геноме имеют одну и ту же эволюционную историю и, соответственно, описываются одним и тем же эволюционным деревом. Но из-за реассортации может оказаться, что история разных генов, попавших в один геном, различается, поскольку вирусные частицы-«родители» имеют разное происхождение. Тогда, например, может оказаться, что сегмент HA сидит на одной ветке, а сегмент NA, взятый из того же генома — совсем на другой ветке дерева. Существуют компьютерные программы, которые ищут реассортации как раз по таким несоответствиям между эволюционными деревьями разных генов. Одна из таких программ называется Graphincompatibility-based Reassortment Finder (GiRaF) [14]. Принцип ее работы заключается в построении для некой выборки секвенированных вирусов отдельных филогенетических деревьев по каждому из восьми сегментов генома. Последующее сравнение этих деревьев позволяет обнаружить ветви, в которых построенные деревья не совпадают друг с другом. Такие ветви указывают на то, что в данный период эволюции вируса произошла реассортация, поскольку иначе деревья бы совпали.
Подобное исследование можно провести с использованием геномов вируса гриппа, опубликованных в свободном доступе. Избрав в качестве объекта штаммы H3N2, можно составить выборку из 1376 сегментированных геномов, а затем сравнить между собой филогенетические деревья для этих вирусов, построенные в отдельности по каждому из геномных сегментов [15].
В результате такого сравнения оказалось, что число реассортаций примерно сопоставимо для разных сегментов: в ходе эволюции гриппа в популяции человека каждая пара сегментов в недавнем прошлом реассортировала около 50 раз.
Эволюционные деревья (а), построенные по разным генам (дерево слева — по гену гемагглютинина, справа — по гену нейраминидазы) одного и того же набора штаммов вируса гриппа [15]. Сравнение деревьев позволяет обнаруживать реассортации — моменты эволюционной истории, когда геномы разных штаммов «перемешивались». Без реассортаций два дерева были бы одинаковыми; из-за реассортаций кажется, что некоторые ветки на одном дереве «отрезали и перепривили», чтобы получилось другое дерево. Соответствующие друг другу «листья» на «перепривитых» ветках соединены линиями разных цветов. На увеличенном фрагменте дерева (б) красным кружком обозначен момент реассортации, зелеными прямоугольниками — следовавшие за ним изменения аминокислотной последовательности. На масштабных линейках указана доля изменившихся нуклеотидов
Последствия реассортаций
После того как ветви, в которых произошли реассортации, были обнаружены, стало возможным оценить их влияние на накопление в сегментах вирусного генома точечных замен. Для этого можно сравнить время, прошедшее между каждой такой заменой и ближайшей предшествующей ей реассортацией, с тем, которое бы ожидалось из компьютерной модели, если бы реассортации не влияли на замены. Проведенный анализ показал, что по крайней мере в пяти из восьми сегментов генома мутации ускоренно накапливаются после реассортации. Наиболее ярко эффект проявился для нейраминидазы и белка PB1. Ускорение аминокислотных замен после реассортаций вирусных геномов указывает на то, что в такие периоды эволюции вируса гриппа прежде всего происходит адаптация белков к новому генетическому окружению. Из-за того, что вирусные белки взаимодействуют между собой, молекулы из разошедшихся штаммов вынуждены какое-то время изменяться, приспосабливаясь друг к другу.
При этом для разных сегментов эффект ускоренного накопления точечных замен длится разное время. Если реассортация затрагивает субъединицу полимеразы, период «притирки» сегментов к новому копирующему белку оказывается сравнительно долгим. В случае нейраминидазы период повышенной скорости замен короче, и большинство дополнительных точечных замен встречается на филогенетических ветвях, которые сразу следуют за той, что отмечена реассортацией.
Интересно, что у нейраминидаз наблюдалось 30 замен, расстояние от которых до ветви, несущей реассортацию, меньше того эволюционного расстояния, на котором мы бы ожидали встретить одну случайную синонимичную замену в гене данного белка. Такой результат свидетельствует о том, что все эти 30 мутаций произошли и закрепились необычайно быстро, и что необходимость быстрой адаптации возникла именно благодаря тому, что соответствующий сегмент генома попал в новое генетическое окружение.
Какие же именно мутации случаются после реассортаций? Чтобы ответить на этот вопрос, можно разделить мутации на разные категории, пользуясь уже имеющимися знаниями о биологии вируса, о структуре его белков и об их эволюции. Например, известно, что некоторые мутации подвержены так называемому положительному отбору. Это значит, что они увеличивают приспособленность данного штамма — возрастает число вирусных частиц-потомков, которые он производит. В результате на такие «полезные» для вируса мутации начинает действовать дарвиновский естественный отбор, и частота их встречаемости со временем повышается. Такой отбор в поверхностных белках вируса гриппа (гемагглютинине и нейраминидазе), как правило, связан с действием иммунной системы хозяина: более приспособленными оказываются такие мутантные частицы, которые эффективнее «маскируются», избегая иммунитета. Места в белках, которые вирус может изменить, чтобы стать незаметным для иммунной системы, и которые поэтому обычно подвержены такому отбору, хорошо известны [16]. Можно было бы ожидать, что эти места также эволюционируют быстрее и после реассортаций. Но оказалось, что ускорение эволюции после реассортаций происходит совсем в других частях белка. По-видимому, это означает, что за взаимодействия белков друг с другом отвечают не те участки, что за их взаимодействие с иммунной системой.
Кроме того, можно изучить параллельные мутации, которые многократно затрагивали одни и те же позиции в уже эволюционно разошедшихся белках. Оказалось, что такие мутации часто случаются и вскоре после реассортаций. Возможно, это означает, что существенная доля параллельных мутаций происходит как ответ на мутации в других белках вируса. Вирус — тесно скоординированная структура, и, возможно, не только реассортации, но и каждое важное эволюционное событие влечет за собой «шлейф» изменений в других местах того же белка или других белков. Взаимодействия между разными генами и между разными местами одного гена называются эпистазом. Пары замен у вируса гриппа, когда вторая замена нейтрализует вредные последствия первой, были известны и ранее [17]. Оказалось, что замены в сайтах, способные уравновешивать изменения в других местах того же белка, быстро возникают также и после реассортаций, по-видимому, «подстраивая» межбелковые взаимодействия после замен в другом белке. Похоже, существуют такие аминокислоты, изменения которых могут нейтрализовать множество различных «поломок» в вирусном геноме.
Реассортация — это резкое эволюционное изменение, которое поначалу может снижать общую приспособленность вируса к условиям окружающей среды и к организму-хозяину. Однако иногда оказывается, что из-за такой перетасовки белков из разных штаммов новая форма патогена оказывается более приспособленной, чем штаммы-предшественники, получая возможность эффективнее распространиться [18]. Похоже, что за коррекцию первичного вредного эффекта от реассортации как раз и отвечают быстро закрепляющиеся адаптивные мутации.
Предсказания, полученные только статистическими методами, — путем анализа последовательностей белков и кодирующих их генов, — конечно, не могут иметь стопроцентную точность. Действительно ли взаимодействуют две определенные аминокислоты, можно проверить экспериментально. Однако каждый белок вируса состоит из сотен аминокислот, так что возможны десятки тысяч разных взаимодействий. Постановка такого числа экспериментов практически неосуществимы. Биоинформатический анализ позволяет расставлять приоритеты: выбирать и анализировать только те аминокислоты, которые участвуют во взаимодействиях, экономя время и силы экспериментаторов. Кроме того, такой подход позволяет понять, насколько взаимодействия, приводящие к вредности реассортаций, распространены на уровне всего генома.
Вопрос этот отнюдь не праздный. Связь между реассортациями и последующим накоплением аминокислотных замен может быть важна для предсказания возникновения новых линий вируса, способных вызвать пандемии. Например, «птичий» грипп (H5N1) — один из вероятных кандидатов на роль агента будущих серьезнейших эпидемий [19, 20]. Существующие штаммы этого подтипа гриппа в настоящее время не передаются между млекопитающими; однако для того чтобы получить такую способность, вирусу необходимы всего лишь пять дополнительных мутаций либо реассортация в сочетании с четырьмя заменами [21, 22]. При этом две из этих мутаций уже распространены среди вирусов H5N1 [23]. Если произойдет реассортация, недостающие замены могут возникнуть быстрее, чем считалось ранее.
Работа выполнена при поддержке Российского фонда фундаментальных исследований (проект 13-04-02098) и Министерства образования и науки Российской Федерации (проект 11.G34.31.0008).
Об авторах
Георгий Александрович Базыкин — кандидат биологических наук, заведующий сектором молекулярной эволюции в Институте проблем передачи информации им. А. А. Харкевича РАН, ведущий научный сотрудник лаборатории эволюционной геномики факультета биоинженерии и биоинформатики МГУ им. М. В. Ломоносова. Занимается изучением различных вопросов биологической эволюции с использованием методов геномики и биоинформатики.
Юрий Эдуардович Стефанов — кандидат биологических наук, научный сотрудник Института молекулярной биологии РАН им. В. А. Энгельгарта и научный консультант студии научного дизайна Visual Science. Область научных интересов — эволюция мобильных генетических элементов, трехмерное компьютерное моделирование вирусных частиц.
Литература
1. Steinhauer D. A., Domingo E., Holland J. J. Lack of evidence for proofreading mechanisms associated with an RNA virus polymerase // Gene. 1992. V. 22. № 2. P. 281–288.
2. Teng Q., Hu T., Li X. et al. Complete genome sequence of an H3N2 avian influenza virus isolated from a live poultry market in Eastern China // J. Virol. 2012. V. 86. № 21. P. 11944. DOI: 10.1128/JVI.02082-12.
3. Carr C. M., Kim P. S. A spring-loaded mechanism for the conformational change of influenza hemagglutinin // Cell. 1993. V. 73. № 4. P. 823–832.
4. Arranz R., Coloma R., Chichуn F. J. et al. The structure of native influenza virion ribonucleoproteins // Science. 2012. V. 338. № 6114. P. 1634–1637. DOI: 10.1126/science.1228172.
5. Kamali A., Holodniy M. Influenza treatment and prophylaxis with neuraminidase inhibitors: a review // Infection and Drug Resistance. 2013. № 6. P. 187–198. DOI: 10.2147/IDR.S36601.
6. Nayak D. P., Hui E. K., Barman S. Assembly and budding of influenza virus // Virus Res. 2004. V. 106. № 2. P. 147–165.
7. Lear J. D. Proton conduction through the M2 protein of the influenza A virus; a quantitative, mechanistic analysis of experimental data // FEBS Lett. 2003. V. 552. № 1. P. 17–22.
8. Hale B. G., Randall R. E., Ortнn J. et al. The multifunctional NS1 protein of influenza A viruses // J. Gen. Virol. 2008. V. 89. № 10. P. 2359–2376. DOI: 10.1099/vir.0.2008/004606-0.
9. Robb N. C, Smith M., Vreede F. T. et al. NS2/NEP protein regulates transcription and replication of the influenza virus RNA genome // J. Gen. Virol. 2009. V. 90. № 6. P. 1398–1407. DOI: 10.1099/vir.0.009639-0.
10. El Zowalaty M. E., Bustin S. A., Husseiny M. I. et al. Avian influenza: virology, diagnosis and surveillance // Future Microbiol. 2013. V. 8. № 9. P. 1209–1227. DOI: 10.2217/fmb.13.81.
11. Kaplan B. S., Webby R. J. The avian and mammalian host range of highly pathogenic avian H5N1 influenza // Virus Res. 2013. V. 178. № 1. P. 3–11. DOI: 10.1016/j.virusres.2013.09.004.
12. Kilbourne E. D. Influenza pandemics of the 20th century // Emerg. Infect. Dis. 2006. V. 12. № 1. P. 9–14.
13. Suzuki Y. A phylogenetic approach to detecting reassortments in viruses with segmented genomes // Gene. 2010. V. 464. № 1–2. P. 11–16. DOI: 10.1016/j.gene.2010.05.002.
14. Nagarajan N., Kingsford C. GiRaF: robust, computational identification of influenza reassortments via graph mining // Nucleic Acids Research. 2011. V. 39. № 6. e34. DOI: 10.1093/nar/gkq1232.
15. Neverov A. D., Lezhnina K. V., Kondrashov A. S., Bazykin G. A. Intrasubtype Reassortments Cause Adaptive Amino Acid Replacements in H3N2 Influenza Genes // PLoS Genet. 2014. V. 10. № 1. e1004037. DOI: 10.1371/journal.pgen.1004037
16. Wolf Y. I., Viboud C., Holmes E. C. et al. Long intervals of stasis punctuated by bursts of positive selection in the seasonal evolution of influenza A virus // Biol. Direct. 2006. V. 1. P. 34.
17. Kryazhimskiy S., Dushoff J., Bazykin G. A. et al. Prevalence of epistasis in the evolution of influenza A surface proteins // PLoS Genet. 2011. V. 7. № 2. e1001301. DOI: 10.1371/journal.pgen.1001301.
18. Li K. S., Guan Y., Wang J. et al. Genesis of a highly pathogenic and potentially pandemic H5N1 influenza virus in eastern Asia // Nature. 2004. V. 430. № 6996. P. 209–213.
19. Ferguson N. M., Fraser C., Donnelly C. A. et al. Public health. Public health risk from the avian H5N1 influenza epidemic // Science. 2004. V. 304. № 5673. P. 968–969.
20. Yong E. Influenza: Five questions on H5N1 // Nature. 2012. V. 486. № 7404. P. 456–458. DOI: 10.1038/486456a.
21. Herfst S., Schrauwen E. J., Linster M. et al. Airborne transmission of influenza A/H5N1 virus between ferrets // Science. 2012. V. 336. № 6088. P. 1534–1541. DOI: 10.1126/science.1213362.
22. Imai M., Watanabe T., Hatta M. et al. Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets // Nature. 2012. V. 486. № 7403. P. 420–428. DOI: 10.1038/nature10831.
23. Russell C. A., Fonville J. M., Brown A. E. et al. The potential for respiratory droplet-transmissible A/H5N1 influenza virus to evolve in a mammalian host // Science. 2012. V. 336. № 6088. P. 1541–1547. DOI: 10.1126/science.1222526.
https://elementy.ru/nauchno-populyarnaya_biblioteka/434482/S...



Наука | Научпоп
7.6K постов78.4K подписчика
Правила сообщества
Основные условия публикации
- Посты должны иметь отношение к науке, актуальным открытиям или жизни научного сообщества и содержать ссылки на авторитетный источник.
- Посты должны по возможности избегать кликбейта и броских фраз, вводящих в заблуждение.
- Научные статьи должны сопровождаться описанием исследования, доступным на популярном уровне. Слишком профессиональный материал может быть отклонён.
- Видеоматериалы должны иметь описание.
- Названия должны отражать суть исследования.
- Если пост содержит материал, оригинал которого написан или снят на иностранном языке, русская версия должна содержать все основные положения.
Не принимаются к публикации
- Точные или урезанные копии журнальных и газетных статей. Посты о последних достижениях науки должны содержать ваш разъясняющий комментарий или представлять обзоры нескольких статей.
- Юмористические посты, представляющие также точные и урезанные копии из популярных источников, цитаты сборников. Научный юмор приветствуется, но должен публиковаться большими порциями, а не набивать рейтинг единичными цитатами огромного сборника.
- Посты с вопросами околонаучного, но базового уровня, просьбы о помощи в решении задач и проведении исследований отправляются в общую ленту. По возможности модерация сообщества даст свой ответ.
Наказывается баном
- Оскорбления, выраженные лично пользователю или категории пользователей.
- Попытки использовать сообщество для рекламы.
- Фальсификация фактов.
- Многократные попытки публикации материалов, не удовлетворяющих правилам.
- Троллинг, флейм.
- Нарушение правил сайта в целом.
Окончательное решение по соответствию поста или комментария правилам принимается модерацией сообщества. Просьбы о разбане и жалобы на модерацию принимает администратор сообщества. Жалобы на администратора принимает и общество Пикабу.